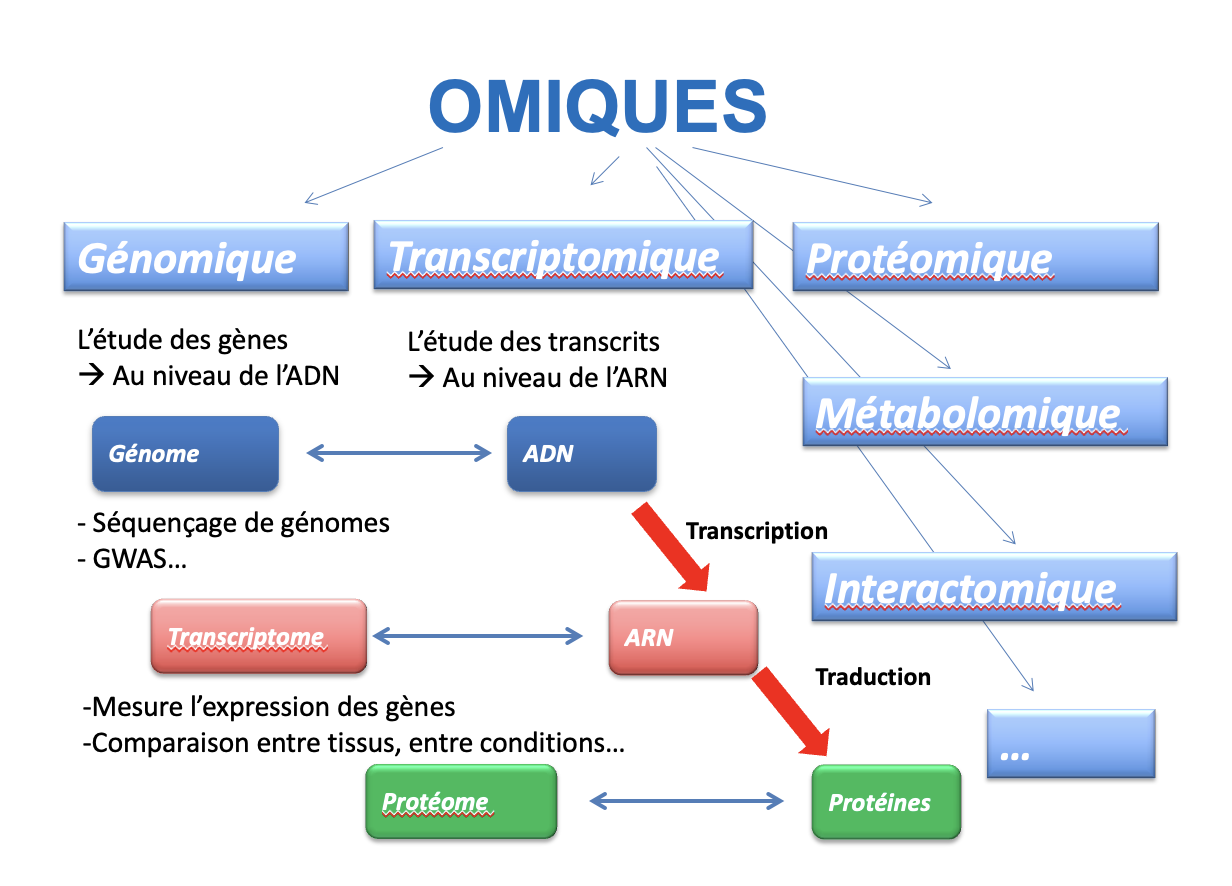
Introduction aux omiques : Evolution des technologies, objectifs, figures classiques: heatmap, boxplot, violin plot, volcanoplot manatthan plot, notion de tests multiples, outils moléculaires pour l'interactomique
Rappel sur les Genome Browser (UCSC/ Ensembl) : analyse des transcrits, Données SNP et OMIM, données ENCODE, séquences répéteés, analyse de conservation, analyse de synténie
Technologie ADN : short reads and long reads NGS, microarray, DNA Game, bioinformatique pour le traitement des données NGS
Transcriptomique : régulation génique, épigénétique, analyse différentielle d'expression, visualisation des reads, applications et leur analyse bionformatique (ChiP-seq ATAC-seq, Hi-C...)
Génomique : assemblage de génome, liaison et déquilibre de liaison, approche candidat, GWAS, eQTL, métagénomique (whole genome and metabarcoding )
Protéomique: MS, MS/MS, identification avec MASCOT, analyse différentielle, interactomique
Métabolomique : identification, MWAS